science
Chronologie de la technologie de séquençage de l’ADN sur la Station spatiale internationale


iss057e000185 (10/8/2018) – Le séquenceur biomoléculaire Best Experiment flotte devant la fenêtre 7 de l’unité de copule. La Terre en arrière-plan. Un séquenceur biomoléculaire cherche à prouver, pour la première fois, que le séquençage de l’ADN est possible dans un vaisseau spatial en orbite autour de la Terre. Un séquenceur d’ADN spatial pourrait identifier les microbes, diagnostiquer des maladies, comprendre la santé des membres d’équipage et peut-être aider à découvrir la vie basée sur l’ADN ailleurs dans le système solaire.
NASA
Les bactéries peuvent être identifiées grâce à leur schéma biologique unique, trouvé dans les molécules d’acide désoxyribonucléique (ADN).
L’ADN se compose de quatre molécules de base qui s’unissent pour coder des instructions relatives à la croissance et au comportement des cellules. La détermination de l’ordre des bases à l’aide du processus de séquençage de l’ADN indique aux chercheurs qui sont les organismes et comment ils pourraient se comporter.
Historiquement, l’équipement nécessaire au séquençage de l’ADN était coûteux, prenait beaucoup de temps et nécessitait une expertise spécialisée pour fonctionner, ce qui limitait son utilisation dans l’espace.
Découvrez comment cette technologie a évolué jusqu’à ce que les chercheurs puissent désormais séquencer l’ADN à bord de la Station spatiale internationale :
Février 1953 – Francis Crick, James Watson et Rosalind Franklin découvrent la structure en double hélice qui constitue l’ADN.
Décembre 1977 – Frederick Sanger développe la première méthode de séquençage de l’ADN pour lire le génome du virus.
Juillet 1995 – Premier séquençage complet du génome bactérien (H. influenzae) à l’aide du séquençage shotgun, qui divise le génome en petites parties séquencées individuellement à l’aide d’une méthode de terminaison de chaîne, puis réassemblées.
Février 2012 – Oxford Nanopore Technologies a lancé le premier séquenceur de nanopores utilisant le séquençage de nouvelle génération (NGS) avec MinION.

Avril 2016 – Dans le cadre de l’étude WetLab-2 de la NASA, l’astronaute de la NASA Jeff Williams a réalisé le premier isolement de l’ARN d’Escherichia coli dans l’espace et a collecté des données sur les niveaux d’expression de l’ARN dans le microbe.
Avril 2016 – L’ADN a été amplifié pour la première fois à bord de la station par l’astronaute de l’ESA Tim Peake à l’aide du premier appareil PCR envoyé à la station par la société miniPCR.


Août 2016 – L’astronaute de la NASA Kate Rubin séquence pour la première fois l’ADN dans l’espace.

Août 2017 – L’astronaute de la NASA Peggy Whitson combine la miniPCR avec MinION pour séquencer et identifier le premier microbe inconnu de la station.

Une démonstration de l’expérience Genes in Space-3 dans le module Node 2. Les expériences Genes in Space-3 démontrent comment le séquençage portable de l’ADN en temps réel peut être utilisé pour examiner l’environnement microbien, diagnostiquer les maladies infectieuses et surveiller la santé de l’équipage à bord. la Station spatiale internationale.

Août 2018 – L’astronaute de la NASA Ricky Arnold présente pour la première fois la technologie d’extraction et de séquençage biomoléculaire (BEST) en utilisant des méthodes indépendantes de la culture pour séquencer l’ADN sur la station à l’aide de la méthode « écouvillonnage vers séquence ». Ce processus accélère le taux de séquençage, ne nécessitant plus le temps et les ressources nécessaires à la croissance des bactéries avant l’analyse.

Mai 2019 – L’astronaute de la NASA Christina Koch effectue la première édition génétique CRISPR-Cas9 sur la station, en utilisant de la levure pour imiter les effets du rayonnement spatial sur l’ADN humain.

31 juillet 2020 – Chris Cassidy, astronaute de la NASA et commandant de l’expédition 63, travaille à l’intérieur du module Harmony de la Station spatiale internationale et sert des échantillons d’ADN microbien à des fins de séquençage et d’identification.

Février 2021 – L’équipage a effectué plus de 800 prélèvements d’échantillons microbiens dans toute la station pour l’expérience 3DMM. Les scientifiques ont utilisé le séquençage de l’ADN et d’autres analyses pour créer la première carte 3D complète des bactéries et des produits bactériens dans toute la station.

3 janvier 2022 – L’astronaute de la NASA et ingénieur de vol de l’Expédition 66, Raja Chari, séquence l’ADN d’échantillons de bactéries à l’aide de l’installation BioMole pour comprendre l’environnement microbien de la Station spatiale internationale.

6 septembre 2023 – L’astronaute de la NASA et ingénieur de vol de l’Expédition 69, Yasmine Moghbeli, fournit des échantillons microbiens pour le séquençage de l’ADN à bord de la Station spatiale internationale.

Astrobiologie, génomique, astrobiologie,

« Spécialiste de la télévision sans vergogne. Pionnier des zombies inconditionnels. Résolveur de problèmes d’une humilité exaspérante. »
science
Le chef de l’Agence spatiale américaine veut parler avec la Chine des débris spatiaux

29 août 2024
Le chef du commandement spatial américain espère que la prochaine fois que la Chine lancera un missile laissant derrière elle des débris spatiaux de longue durée, Pékin avertira Washington à l’avance, plutôt que de laisser les États-Unis découvrir par eux-mêmes le chaos orbital.
Parlez dans L’efficacité du Mitchell Institute for Aerospace Studies Lors d’une réunion à la base spatiale Peterson, au Colorado, le 28 août, le général Stephen N. Whiting a souligné deux incidents récents impliquant des débris spatiaux chinois comme étant une source de préoccupation et la nécessité d’améliorer la communication à l’avenir.
« Nous venons de voir le lancement de leur version de Constellation du soleil « Cela a laissé plus de 300 débris en orbite – une fusée Longue Marche 6A », a déclaré Whiting. « Il y a moins de deux ans, ils avaient une autre fusée, qui a mis plus de 500 débris à longue durée de vie… J’espère que la prochaine fois. que « Dans un missile comme celui-ci, il laisse beaucoup de débris. Ce ne sont pas nos capteurs qui détectent cela en premier, mais nous obtenons des communications qui nous aident à comprendre cela, tout comme nous communiquons avec les autres. »
L’incident le plus récent impliquant une fusée Longue Marche 6A s’est produit au début du mois, lorsque le lanceur transportait les 18 premiers satellites d’une constellation de communications prévue pour rivaliser avec Starlink. La fusée s’est brisée en orbite terrestre basse (LEO) quelques jours plus tard, répandant des débris et suscitant des inquiétudes parmi les experts. La société privée de suivi spatial a rapporté que la désintégration pourrait produire plus de 10… 900 épaves shrapnel.

Whiting a noté que les débris provenaient de l’étage supérieur de la fusée après le lancement des satellites, indiquant que la mission était « généralement réussie ». Cependant, à des altitudes plus élevées, les débris resteront en orbite plus longtemps.
« Nous ne voulons certainement pas voir ce genre de débris », a ajouté Whiting.
Les débris se trouvent généralement sur des orbites inférieures à 600 km (373 miles). Il revient sur Terre après quelques annéesÀ une altitude de 800 km, sa décomposition peut prendre des siècles. Avec de plus en plus de satellites en orbite terrestre basse et des débris persistants provenant de lancements peu judicieux, La probabilité de collisions continue d’augmenter.
Selon le général à la retraite Kevin Shelton, directeur du Centre d’excellence sur l’énergie spatiale du Mitchell Institute, les États-Unis ont déjà eu des problèmes similaires avec des débris à haute altitude, mais ont commencé à évacuer le carburant et les gaz des étages de fusée avant d’entrer en orbite. Cette pratique réduisait les débris et le risque de désintégration, et la Russie l’adopta peu après. Whiting a déclaré qu’on ne savait pas actuellement si la Chine utilisait cette méthode.
« Depuis des décennies, les États-Unis s’intéressent tellement à l’espace que nous avons mis la grande majorité de nos données de suivi à la disposition du monde entier », a déclaré Whiting. « Chaque jour, nous analysons tous les satellites actifs à la recherche de tous ces débris, et nous en informons tout le monde, y compris les Chinois et les Russes… parce que nous ne voulons pas que les satellites heurtent des débris et laissent derrière eux d’autres débris. »
Le développement rapide des capacités spatiales et l’augmentation significative des déploiements de satellites par la Chine et la Russie restent une préoccupation majeure quant à la manière dont les États-Unis abordent le domaine spatial. Chef adjoint des opérations spatiales, le général Michael A. Gotlin a souligné que les récentes mesures prises par ces pays prouvent leur intention d’opérer de manière dangereuse dans ce domaine.
« Ils créent beaucoup de débris et d’orbites que nous devons contourner, ou ils mettent en danger des choses comme la Station spatiale internationale », a déclaré Gotlin lors du Sommet AFCEA/INSA sur le renseignement et la sécurité nationale à Rockville, Maryland, le 28 août. Il a ajouté : « Ils ne se soucient même pas de la sécurité des astronautes. Si ce n’est pas dangereux et non professionnel, je ne sais pas ce que c’est. »
En novembre 2021, la Russie a procédé à un test de missile antisatellite, aboutissant à la création d’un Grande quantité de débris En orbite terrestre basse, ce qui présente un danger pour la Station spatiale internationale et incite l’équipage à prendre des mesures de précaution. En outre, Moscou a également été témoin Une série de fuites de liquide de refroidissement Ces dernières années, la Chine a lancé son propre vaisseau spatial. Même s’il n’y a pas de négociations prévues avec la Russie sur le développement spatial, les espoirs sont grands d’une communication plus active avec Pékin sur les alertes spatiales.
« Nous donnons ces avis aux Chinois, et au cours de l’année dernière, nous avons vu à plusieurs reprises qu’ils nous ont donné quelques avis en retour, et je pense que c’est une chose positive. Nous n’avons aucune discussion. prévu avec la Russie », a déclaré Whiting.

« Spécialiste de la télévision sans vergogne. Pionnier des zombies inconditionnels. Résolveur de problèmes d’une humilité exaspérante. »
science
À la recherche de pierres précieuses : caractérisation de six planètes géantes en orbite autour de naines froides
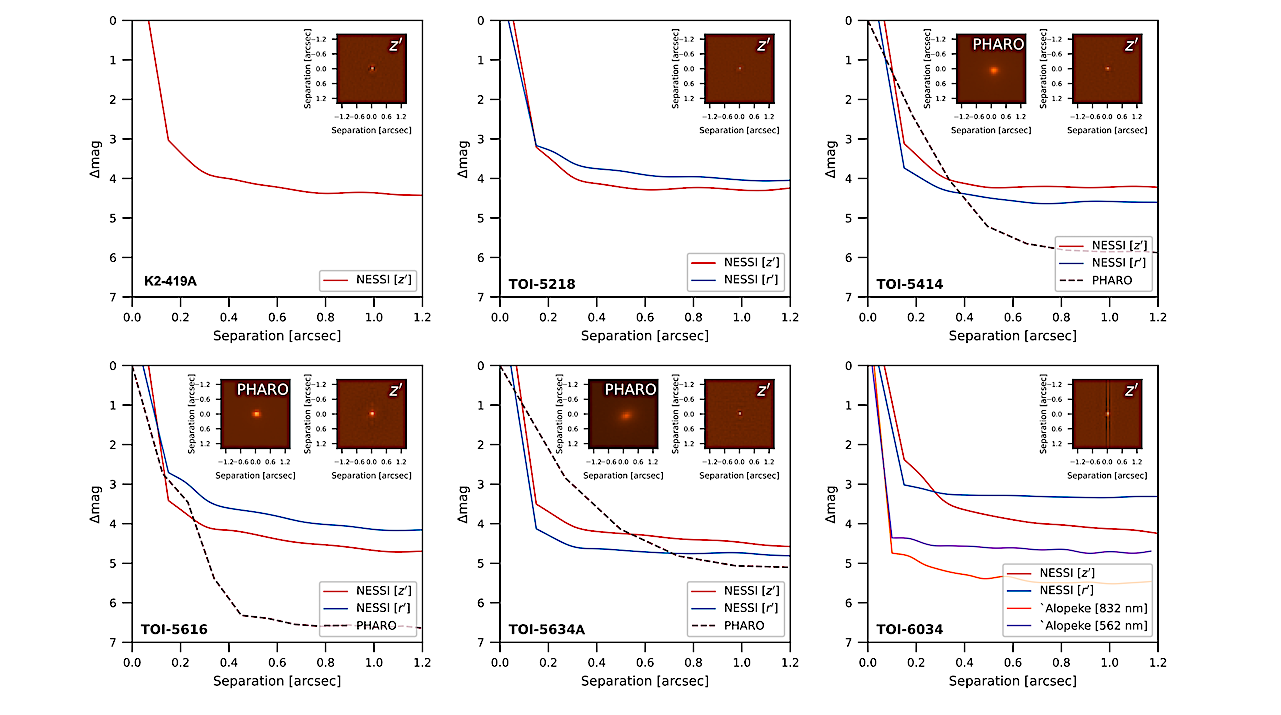

Données d’imagerie à contraste élevé pour toutes les cibles. Dans chaque panneau, nous montrons la limite de contraste de 5σ atteinte en fonction de la séparation angulaire de l’étoile hôte pour chaque ensemble de données à contraste élevé. Nous traçons également des cachets postaux de 1,4″ × 1,4″ d’images NESSI reconstruites en bande z (encadré à droite dans chaque panneau) pour toutes les cibles et des images AO (encarts à gauche) pour TOI-5414, TOI-5616, TOI-5634A et TOI-6034. — astro-ph.EP
Les exoplanètes géantes transitant autour d’étoiles naines de type M (GEMS) sont rares, en raison de la faible masse de leurs étoiles hôtes. Cependant, la couverture de l’ensemble du ciel par TESS a permis d’en détecter un nombre croissant pour permettre des enquêtes statistiques telles que le GEMS Search Survey.
Dans le cadre de cet effort, nous décrivons les observations de six planètes géantes en transit, qui incluent des mesures de masse précises pour deux GEMS (K2-419Ab, TOI-6034b) et une validation statistique de quatre systèmes, qui incluent une vérification et des limites de masse supérieures pour trois d’entre elles. (TOI-5218b, TOI-6034b). 5616b, TOI-5634Ab), tandis que le quatrième système – TOI-5414b – est classé comme « planète potentielle ».
Nos observations incluent les vitesses radiales du Habitable Zone Planet Finder sur le télescope Hobby-Eberly et de l’observatoire Maroon-X sur Gemini-North, ainsi que la photométrie et l’imagerie à contraste élevé provenant de plusieurs installations au sol. En plus de la photométrie TESS, K2-419Ab a également été observé et validé statistiquement dans le cadre de la mission K2 au cours des campagnes 5 et 18, qui fournit des contraintes orbitales et planétaires précises malgré la faible luminosité de l’étoile hôte et la longue période orbitale d’environ 20,4 jours.
Avec une température d’équilibre de seulement 380 K, K2-419Ab est l’une des planètes en transit les plus froides et les mieux caractérisées connues. TOI-6034 a un compagnon tardif de type F à environ 40 secondes d’arc, ce qui en fait la première étoile hôte GEMS à avoir un ancien compagnon binaire sur la séquence principale. Ces confirmations s’ajoutent au petit échantillon existant de planètes en transit GEMS confirmées.
Shubham Kanodia, Arvind F. Gupta, Caleb I. Canas, Lea Marta Bernabo, Varghese Reggie, T. Hahn, Madison Brady, Andreas Seyfart, William D. Cochrane, Nydia Morrell, Ritvik Basant, Jacob Bean et Chad F. Bender, Zoé L. De Bors, Alison Perella, Alexina Birkholz, Nina Brown, Franklin Chapman, David R. Ciardi, Catherine A. Clark, Ethan J. Cotter, Scott A. Diddams, Samuel Halverson, Susan Hawley, Leslie Hebb, Ray Holcomb, Steve B. Howell, Henry A. Kobolnicki, Adam F. Kowalski, Alexander Larsen, Jessica Libby Roberts, Andrea S. J. Lin, Michael B. Lund, Raphael Locke, Andrew Munson, Joe B. Ninan, Brooke A. Parker, Nishka Patel, Michael Rudrak, Gabrielle Ross, Arpita Roy, Christian Schwab, Jomundur Stefansson, Aubrey Thoms, Andrew Vanderberg
Commentaires : Accepté dans AJ
Sujets : Astrophysique terrestre et planétaire (astro-ph.EP)
Citer ce qui suit : arXiv:2408.14694 [astro-ph.EP] (ou arXiv :2408.14694v1 [astro-ph.EP] (pour cette version)
https://doi.org/10.48550/arXiv.2408.14694
Concentrez-vous pour en savoir plus
Date de publication
De : Shubham Kanodia
[v1] Lundi 26 août 2024, 23:47:24 UTC (5 169 Ko)
https://arxiv.org/abs/2408.14694
Astrobiologie

« Spécialiste de la télévision sans vergogne. Pionnier des zombies inconditionnels. Résolveur de problèmes d’une humilité exaspérante. »
science
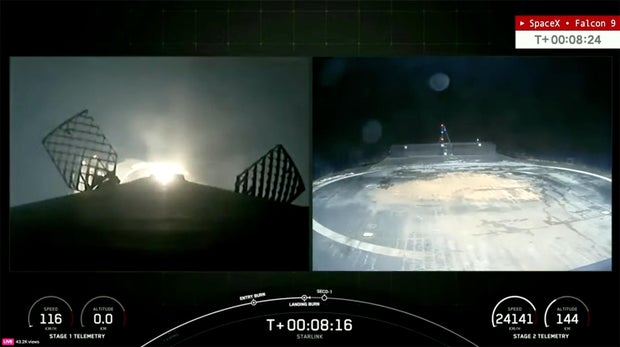
La Federal Aviation Administration des États-Unis a immobilisé les fusées Falcon 9 de SpaceX dans l’attente d’une enquête sur un rare accident d’atterrissage au large des côtes.

La Federal Aviation Administration a immobilisé les fusées Falcon 9 de SpaceX en attendant une enquête visant à déterminer pourquoi le propulseur du premier étage s’est arrêté. Collision avec un bateau de débarquement tôt mercredi après avoir contribué au lancement d’un autre lot de satellites Internet Starlink.
après Se lever Après avoir reporté mardi soir le lancement du vaisseau spatial avec équipage Polaris Dawn en raison de prévisions météorologiques à long terme défavorables, SpaceX a continué à travailler sur le premier des lancements consécutifs de satellites Starlink, un depuis la Floride et un depuis la Californie.
Mais le deuxième vol a été annulé après que le premier étage utilisé lors du lancement en Floride s’est brisé et est tombé dans l’océan Atlantique alors qu’il tentait d’atterrir sur un drone SpaceX stationné à des centaines de kilomètres au nord-est de Cap Canaveral.
EspaceX
La FAA a déclaré qu’elle ordonnerait une enquête, immobilisant efficacement les fusées Falcon 9 de SpaceX – y compris la fusée Polaris Dawn – jusqu’à ce que l’enquête soit terminée et que les mesures correctives soient approuvées.
« Le retour en vol de la fusée Falcon 9 dépend de la détermination par la FAA que tout système, processus ou procédure lié à l’anomalie n’a pas d’impact sur la sécurité publique », a déclaré la FAA dans un communiqué.
« En outre, SpaceX devra peut-être demander et obtenir l’approbation de la FAA pour modifier sa licence qui inclut des actions correctives et satisfaire à toutes les autres exigences de licence », a ajouté l’agence.
Mardi soir, SpaceX a reporté un lancement prévu mercredi Mission Aube PolarisLe lancement d’un vol commercial comprenant la première sortie dans l’espace par une organisation non gouvernementale a été reporté à vendredi au plus tôt en raison des conditions météorologiques attendues à la fin de la mission. Le lancement a été suspendu indéfiniment dans l’attente d’une enquête sur l’accident à l’atterrissage.
L’échec de l’atterrissage a mis fin à une séquence de 267 récupérations consécutives réussies de boosters remontant à février 2021. Cependant, le deuxième étage de la fusée Falcon 9 a réussi à transporter 21 satellites Starlink sur leur orbite prévue.
L’atterrissage du premier étage semblait normal jusqu’au moment de l’atterrissage, lorsque plus de flammes que d’habitude sont apparues autour de la base de la fusée à l’approche du pont de la fusée. L’une des jambes d’atterrissage s’est effondrée immédiatement après l’atterrissage et la fusée d’appoint, masquée par le feu et la fumée, s’est renversée par-dessus le côté de la péniche de débarquement dans l’océan Atlantique.
EspaceX
EspaceX
EspaceX
« Après une ascension réussie, le premier étage d’une fusée Falcon 9 s’est retourné après son atterrissage sur le vaisseau spatial sans pilote ‘Zero Gravity' », SpaceX Il a dit sur les réseaux sociaux« Les équipes évaluent les données de vol et l’état du missile. »
Il s’agissait du 23e premier étage de la fusée B1062, qui s’est avéré être son dernier lancement et atterrissage, un nouveau record de réutilisabilité. SpaceX autorise les premiers étages de la fusée Falcon 9 pour un maximum de 40 vols par étage.
Peu de temps après le déploiement des satellites Starlink en Floride, la société a annulé le lancement en Californie, qui était prévu à 5 h 58 HAE, pour donner aux ingénieurs plus de temps pour examiner la télémétrie et les séquences vidéo, à la recherche de tout signe de problème. affecter d’autres missiles.
« Retrait de notre deuxième lancement @Starlink la nuit pour donner à l’équipe le temps d’examiner les données d’atterrissage du booster du lancement précédent », a déclaré SpaceX. Il a dit« Une nouvelle date de lancement cible sera partagée une fois disponible. »

« Spécialiste de la télévision sans vergogne. Pionnier des zombies inconditionnels. Résolveur de problèmes d’une humilité exaspérante. »
-
entertainment4 ans ago
Découvrez les tendances homme de l’été 2022
-
Top News4 ans ago
Festival international du film de Melbourne 2022
-
World1 an ago
Aucun « échange de vues préalable » sur la nomination de McGrath avec Ursula von der Leyen, déclare Martin – Irish Times
-
science4 ans ago
Écoutez le « son » d’un vaisseau spatial survolant Vénus
-
science4 ans ago
Les météorites qui composent la Terre se sont peut-être formées dans le système solaire externe
-
Tech4 ans ago
F-Zero X arrive sur Nintendo Switch Online avec le multijoueur en ligne • Eurogamer.net
-
Top News4 ans ago
Comment parier sur le basket : tous les conseils pour continuer et s’amuser
-
Tech3 ans ago
Voici comment Microsoft espère injecter ChatGPT dans toutes vos applications et bots via Azure • The Register